服务价格:
服务分类:服务分类
所用仪器:
依据标准号:
依据标准号:
数据归一化分析
数据的完整性和准确性是后续获得具有统计学和生物学意义的分析结果的必要 条件。在确保实验设计的合理性和实验数据的准确性的基础上,我们首先对数据 的完整性进行检查,对缺失值进行删除或者补充,删除极值,并对数据进行样本 间和代谢物间的归一化处理,以确保各样本之间和代谢物之间可平行比较。 按4.4项下流程对数据进行处理。原始数据中,缺失值超过 50%的代谢物将被去除,不参与后续分析;对代谢物的 表达量进行对数转换并利用 Autoscaling 方法( Mean-centered and divided by the standard deviation of each variable)进行归一化处理。下图显示了正离子模式数据经归一化处理前后的分布情况,结果表明数据经归一化处理后基本呈正态分布。
 归一化前后的样本
归一化前后的样本
主成分分析(PCA Analysis)
主成分分析( Principal Component Analysis, PCA) 是将原本鉴定到的所有代谢物重新线性组合,形成一组新的综合变量,同时根据所分析的问题从中选取几个(通常 2-3 个)综合变量,使它们尽可能多地反映原有变量的信息,从而达到降维的目的。同时,对代谢物进行主成分分析还能从总体上反应组间和组内的变异度。1)总体样本 PCA 分析 采用 PCA 的方法观察所有各组样本之间的总体分布趋势,找出可能存在的离散样本,综合考虑各种因素(样品数,样品珍贵程度,离散程度)决定离散点的除去与否。所有样本 PCA 得分图见下图(对样本进行两两分析的PCA得分图)。
 假手术组和模型组的主成分分析得分图
假手术组和模型组的主成分分析得分图
偏最小二乘判别分析(PLS-DA)/正交偏最小二乘判别分析(OPLS-DA)
不同于主成分分析( PCA)法,偏最小二乘判别分析( Partial Least Squares Discrimination Analysis, PLS-DA)或正交偏最小二乘判别分析( Orthogonal PLS-DA,OPLS-DA)是一种有监督的判别分析统计方法。该方法运用偏最小二乘回归建立代谢物表达量与样品类别之间的关系模型,来实现对样品类别的预测。分别建立两两分组比较的PLS-DA模型(图1)或OPLS-DA模型(图2),模型得到的参数评价会以表格形式提供。其中R2X和R2Y分别表示所建模型对X和Y矩阵的解释率,Q2标示模型的预测能力,理论上R2、Q2数值越接近1说明模型越好,越低说明模型的拟合准确性越差,通常情况下,R2、Q2高于0.5 (50%)较好,高于0.4即可接受,且两者差值不应过大。临床样本由于个体差异大,不可控,尤其大样本时,R2、Q2大小为0.2左右亦可。同时通过计算变量投影重要度( Variable Importance for the Projection, VIP)来衡量各代谢物的表达模式对各组样本分类判别的影响强度和解释能力, 从而辅助标志代谢物的筛选(通常以 VIP值 > 1.0 作为筛选标准)(图4)。
外部验证:为防止模型过拟合,采用七次循环交互验证和200次响应排序检验的方法来考察PLS-DA模型的质量,R2Y和Q2Y 直线的斜率越接近水平直线,模型越有可能过拟合, Q2小于零说明该模型预测能力高。
在PLS-DA图中,Sham组和Model组有明显区分,并且Q2分别等于X和X说明这两个预测模型的预测能力。
下图则是对PLS-DA模型(c)的检验,直线的斜率大,Q2的截距为X,说明PLS-DA模型没有过拟合。
 假手术组和模型组的PLS-DA 得分图
假手术组和模型组的PLS-DA 得分图
 假手术组和模型组的OPLS-DA模型
假手术组和模型组的OPLS-DA模型
 PLS-DA模型的排列检验图
PLS-DA模型的排列检验图
 假手术组和模型组的PLS-DA 模型载荷图,红框所圈的点为VIP>1的代谢产物
假手术组和模型组的PLS-DA 模型载荷图,红框所圈的点为VIP>1的代谢产物
 模型参数评价表
模型参数评价表
单变量统计分析
在进行两组样本间的差异代 谢物分析时,常用的单变量分析方法包括变异倍数分析( Fold Change Analysis, FC Analysis)、 T 检验,以及综合前两种分析方法的火山图( Volcano Plot)。利用 单变量分析可以直观地显示两样本间代谢物变化的显著性,从而帮助我们筛选潜 在的标志代谢物(通常以 FC > 2.0 且 P value < 0.05 作为筛选标准)。
下图 显示了数据的火山图, 图中枚红色点为 FC > 2.0 且 P value < 0.05 的代谢物,即单变量统计分析筛选的差异代谢物。 其余各组的火山图会以附件形式给出。
 数据结果的火山图( Volcano Plot),绿、红色点为显著性差异代谢物(P value < 0.05)
数据结果的火山图( Volcano Plot),绿、红色点为显著性差异代谢物(P value < 0.05)
显著性差异代谢物分析
选择同时有多维统计分析筛选标准( VIP>1)和单变量统计分析筛选标准 ( FC > 2.0 且 P value < 0.05) 的代谢物作为具有显著性差异的代谢物(图12)。鉴定出的显著性差异代谢物会以表格形式给出。

显著性差异的代谢物
 差异代谢物列表
差异代谢物列表
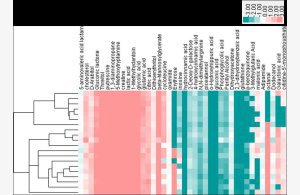
差异代谢产物聚类分析(Hierarchical Clustering)
为了评价候选代谢物的合理性,同时更全面直观地显示样本之间的关系以及代谢 物在不同样本中的表达模式差异,我们利用定性的显著性差异代谢物的表达量对 各组样本进行层次聚类(Hierarchical Clustering),从而辅助我们准确地筛选标志 代谢物,并对相关代谢过程的改变进行研究。 一般来说,当筛选的候选代谢物合理且准确时,同组样本能够通过聚类出现在同 一簇( Cluster)中。同时,聚在同一簇内的代谢物具有相似的表达模式,可能在代谢过程中处于较为接近的反应步骤中。 下图显示了显著性差异代谢物层次聚类结果。
 显著性差异代谢物层次聚类结果
显著性差异代谢物层次聚类结果
差异代谢产物通路分析
将得到的差异代谢物使用MBRole进行代谢通路富集,使用KEGG数据库做为背景,进行相关 通路分析。 选取同物种的所有代谢物作为背景,选取 P value < 0.05的代谢通路。图1和2为我们可提供的两种代谢通路图,富集的代谢通路以及相关T检验分析结果会以表格的形式给出。
 差异代谢物通路图
差异代谢物通路图
 差异代谢物通路图
差异代谢物通路图
| 合同甲方 | 送样单号 | 合同编号 | 评价 | 评语 | 评价日期 |
免费发布咨询信息!
与实验室管理人员面对面沟通。